药物代谢酶的定量技术
药物代谢酶的定量技术
吕雅瑶
药物在体内的吸收、分布、代谢和排泄(ADME)过程受遗传、种族、性别、年龄等诸多因素的影响。其中,药物代谢酶的多态性直接影响药物的代谢动力学过程,进而影响药物疗效。例如,绝大多数抗肿瘤药的治疗指数受代谢酶多态性的影响较大1。因此,药物代谢酶的定量研究对合理用药有重要的指导意义。
肝脏是药物代谢酶分布的主要器官,小肠上皮细胞、肾脏和皮肤中也有分布1。细胞色素P450(CYP450)和尿苷二磷酸葡萄糖醛酸基转移酶(UGT)是两个重要的药物代谢酶家族,介导药物和其它化学物质在体内的转化。
CYP450是一类含有亚铁血红素的药物代谢酶超级家族,包括多个CYP家族,根据氨基酸序列的相似性又可以分为多个亚家族,目前已在人体发现并命名了17种CYP亚家族。CYP450参与甾体、脂质、维生素等的生物合成和降解,催化各种药物的氧化、还原和水解反应(I相反应)。CYP家族中的CYP1、CYP2和CYP3三种药物代谢酶参与了约80%药物的氧化反应以及约50%常用药物的清除反应。不同个体的药物代谢酶含量及活性普遍存在差异且差异往往很大,因而经该类药物代谢物代谢的药物个体间的药效和副反应也存在差异。
UGT酶是一个连接酶家族,能介导内源和外源性化学物质的代谢。UGT酶通过催化尿苷二磷酸葡萄糖醛酸(UDPGA)的葡萄糖醛酸和疏水性底物结合,增加底物极性从而促使底物排出体外(II相反应)。UGT酶按照序列相似性分为两个家族:UGT1和UGT2,这两个家族可以划分为UGT1A、UGT2A和UGT2B三个亚家族。每种UGT酶表现出一定的底物特异性,例如UGT1A1,UGT1A3,UGT1A4,UGT1A6,UGT1A9,UGT2B7,UGT2B10和UGT2B15主要负责肝脏中的药物的葡萄糖醛酸化,而UGT1A10负责肠道中药物的提前清除。
定量药物代谢酶常用的技术有免疫和质谱两种。免疫法是蛋白定量的传统方法,如蛋白免疫印迹法(Western blot)和酶联免疫吸附测定(ELISA)等。和Western blot相比,ELISA的灵敏度更高、线性范围更宽。但是总体而言,免疫法的可靠性差,以测定一个样本的CYP酶含量为例,商品化的标准品中的活性蛋白含量已知,通常假设标准品和样本中的活性蛋白比例相同,再根据标准品中的蛋白含量来测定样本中的CYP酶含量。但是Perrett的研究1证明,标准品和样本中有活性的酶含量往往相差很大,以CYP标准品来定量和以纯化的CYP定量同一个样本相比,前者结果显著低于后者,故定量结果会产生很大误差。另外,免疫测定法不可避免地要使用目标蛋白的抗体,一种蛋白的抗体有可能和它的同分异构体结合而影响定量的准确性。
基于质谱的定量方法不仅可以同时鉴定和定量目标酶,还可以大大提高定量的灵敏度和选择性,并且不需要使用抗体。目前已经逐渐成为定量药物代谢酶的主流方法。
3.1靶向蛋白质组定量技术
选择性反应监测(SRM,或多反应监测,MRM)是使用最广泛的靶向定量方法,被称作蛋白定量的“金标准”。使用SRM技术,需首先选定目标蛋白的特异性肽段和相应的子离子作为定量离子,以三重四极杆(QqQ)质谱对目标蛋白组定量。三重四极杆的Q1识别并选择母离子,在Q2中以氩气或者氮气为碰撞气体将母离子打碎并由Q3选择子离子并最终到达检测器。SRM技术被大量应用于临床诊断以及药物代谢酶的定量研究中。SRM具有高重现性、高选择性和高灵敏度的优点,但也存在一些缺陷,例如,预选定量的母子离子对需要耗费大量工作,定量通量还有很大的提升空间等。Kiyonami等2提出的iSRM(intelligent SRM)在一定程度上提高了SRM的通量和准确性。他们将肽段的一部分子离子用于定量(通常选2-3个),另一部分子离子用于定性,在定量离子的强度达到阈值后再扫描定性子离子,这样能增大定量结果的准确性。由于对每个肽段定性子离子监测时间减少,节省的时间可以用于监测更多的肽段,因此定量通量增大。
近年来随着质谱仪器的迅速发展,轨道离子阱(Orbitrap)、飞行时间(TOF)质谱仪的分辨率、灵敏度和质量准确度都有极大提升,这使得蛋白序列鉴定、翻译后修饰的位点鉴定以及定量的准确度都有了很大的提高。平行反应监测(PRM)和高分辨-多反应监测技术(HR-MRM)技术应运而生,它们分别使用QqOrbitrap和QqTOF为质量分析器。与三重四极杆质谱仪相比,三重四极杆的Q3被替换为高分辨的Orbitrap或TOF;离子扫描时,母离子被打碎后,PRM或HR-MRM同时监测所有子离子,其原理如图1所示。PRM具有以下优势:1、监测所有子离子图谱来匹配母离子,确保了母离子特异性;2、高分辨监测子离子,选择性高,可以有效排除生物样本中复杂基质的干扰;3、无需提前优化定量的子离子,简化方法开发过程。Jian Shi等3用PRM方法测定人的肝脏和HepG2、Hep3B、HuH7三种细胞系中药物代谢酶的表达含量,结果表明,在这三种细胞系中的含量都小于肝脏且不同酶的表达量之间有很大差异。除了肝脏,PRM也常被用在小肠、肾脏等的药物代谢酶定量中4。
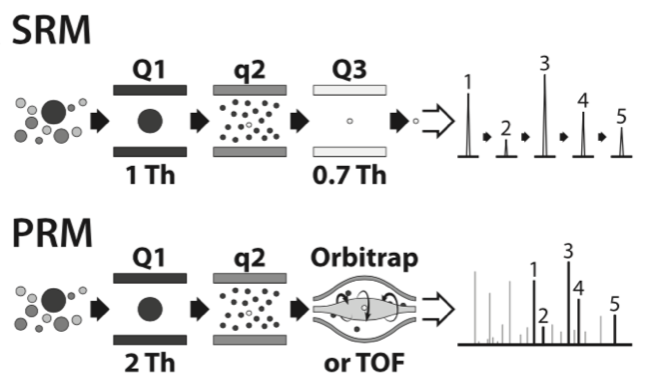
Fig. 1 Schematic diagram of SRM and PRM.
3.2非靶向蛋白质组定量技术
采用数据依赖(DDA)的非靶向蛋白质组技术旨在鉴定更多的蛋白。一级质谱扫描(MS1)提供色谱峰的峰面积或母离子的信号强度,根据MS1图的信息可以定量相应的肽段。每个运行周期,MS1扫描强度较高的一些离子将被选择进行二级碎裂,二级质谱扫描(MS2)可以确定这些肽段的序列,这个过程即DDA。Lane等6研究3,5-二氯-2-[4-[(3,5-二氯-2-吡啶基)氧基]苯氧基]吡啶(TCPOBOP)对CYP450浓度的影响,将小鼠以TCPOBOP处理后取其肝脏蛋白并取正常小鼠的肝脏蛋白做对照,分别用18O和16O标记样本中的蛋白后,用DDA相对定量其中的药物代谢酶含量,共鉴定到17种CYP并定量了其中的16种。研究使用的定量方法操作简单、通量高,可以被广泛应用于评价药物对药物代谢酶含量的影响研究。DDA方法具有通量高、样本前处理简单的优点,但是也存在灵敏度和重现性低的缺点。
数据非依赖采集模式(DIA)将质谱扫描的质量范围划分为多个小窗口,依次扫描并碎裂每个小窗口的所有母离子,用高分辨率的质量分析器Orbitrap或TOF分析这些子离子并利用子离子的信号强度进行定量分析。与DDA不同,DIA采集所有肽段碎裂后的全部子离子,不会造成大量MS1信息遗漏。DIA技术结合了DDA高通量和SRM灵敏度和重现性高的优点,其中一个重要应用SWATH-MS,原理示意图见图2。MS1扫描范围m/z为400-1200 Da,将扫描范围划分为间隔25 Da的多个连续窗口,每个窗口扫描时间为100 ms,一个扫描循环为3.3 s。在一个循环中,首先经过一次MS1扫描,再依次扫描每个窗口内的子离子,记录所有的MS2图。Jamwal等8用QTOFMS测定78例人肝微粒体中的药物代谢酶含量,DDA模式下鉴定到了1,855种蛋白和25,601种肽段并建立谱图库,在SWATH模式下对10种主要的CYP相对定量。
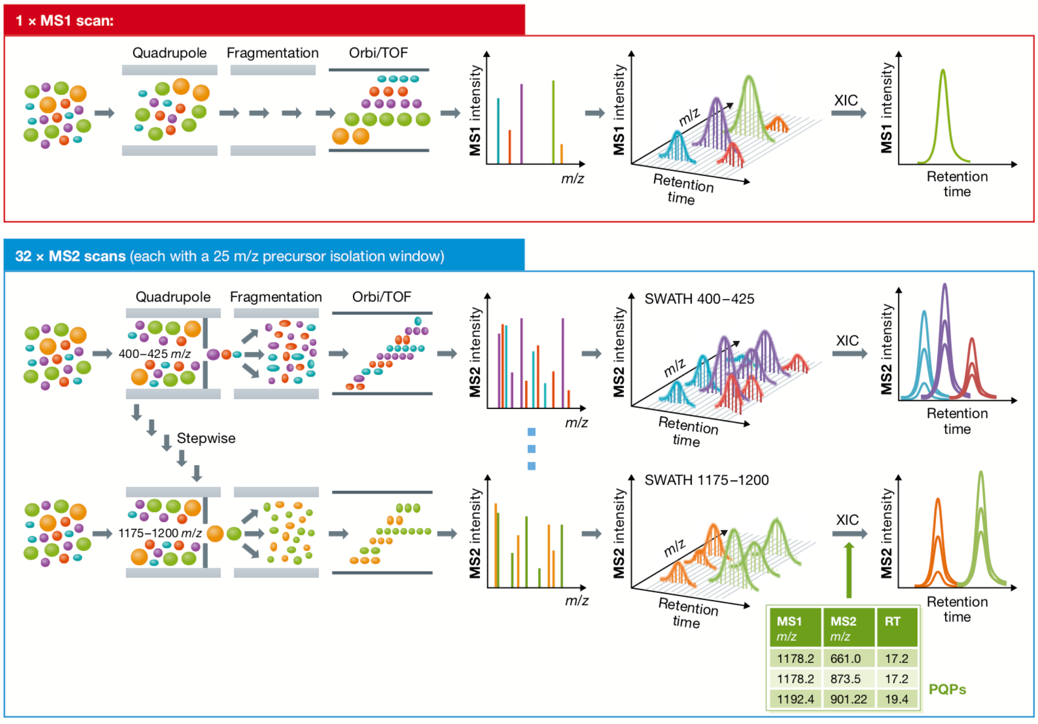
Fig. 2 Schematic diagram of SWATH-MS.
参考文献
1. Perrett H, Barter Z, Jones B, Yamazaki H, Tucker G, Rostami-Hodjegan A. Disparity in holoprotein/apoprotein ratios of different standards used for immunoquantification of hepatic cytochrome P450 enzymes. Drug metabolism and disposition. 2007;35(10):1733-1736.
2. Kiyonami R, Schoen A, Prakash A, et al. Increased selectivity, analytical precision, and throughput in targeted proteomics. Molecular & Cellular Proteomics. 2011;10(2)
3. Shi J, Wang X, Zhu H-J. Comparison of expression profiles of drug-metabolizing enzymes between human liver and the hepatic cell lines HepG2, Hep3B, and HuH7: a parallel reaction monitoring (PRM)-based targeted proteomics study. The FASEB Journal. 2017;31(1_supplement):668.9-668.9.
4. Mimche SM, Lee C-m, Liu KH, et al. A non-lethal malarial infection results in reduced drug metabolizing enzyme expression and drug clearance in mice. Malaria Journal. 2019;18(1)
5. Peterson AC, Russell JD, Bailey DJ, Westphall MS, Coon JJ. Parallel reaction monitoring for high resolution and high mass accuracy quantitative, targeted proteomics. Molecular & Cellular Proteomics. 2012;11(11):1475-1488.
6. Lane CS, Wang Y, Betts R, Griffiths WJ, Patterson LH. Comparative cytochrome P450 proteomics in the livers of immunodeficient mice using 18O stable isotope labeling. Molecular & Cellular Proteomics. 2007;6(6):953-962.
7. Jenkins RE, Kitteringham NR, Hunter CL, et al. Relative and absolute quantitative expression profiling of cytochromes P450 using isotope‐coded affinity tags. Proteomics. 2006;6(6):1934-1947.
8. Jamwal R, Barlock BJ, Adusumalli S, Ogasawara K, Simons BL, Akhlaghi F. Multiplex and label-free relative quantification approach for studying protein abundance of drug metabolizing enzymes in human liver microsomes using SWATH-MS. Journal of proteome research. 2017;16(11):4134-4143.
9. Shi J, Piekos SC, Chen L, Jing X, Zhong X-B, Zhu H-J. Short-and Long-term Effects of Phenytoin Exposure on the Liver Proteome of Neonatal and Adult Mice Using SWATH-MS Technology. The FASEB Journal. 2018;32(1_supplement):563.2-563.2.
10. Ludwig C, Gillet L, Rosenberger G, Amon S, Collins BC, Aebersold R. Data‐independent acquisition‐based SWATH‐MS for quantitative proteomics: a tutorial. Molecular systems biology. 2018;14(8)







